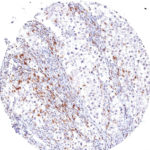
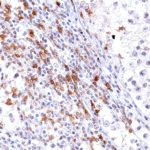
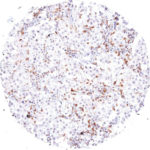
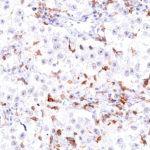
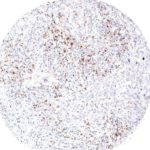
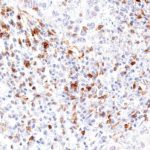
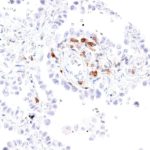
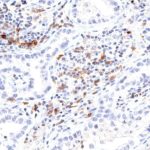
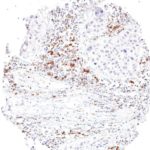
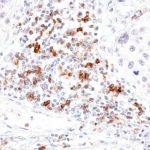
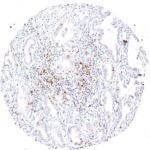
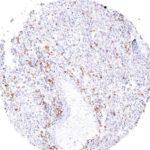
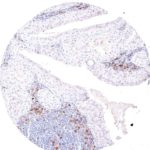
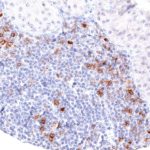
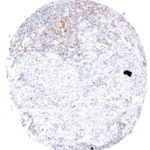
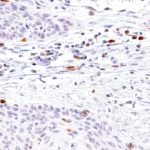
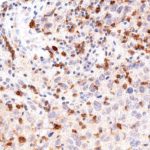
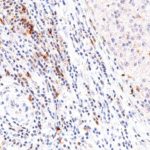
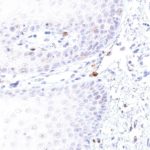
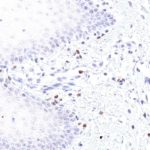
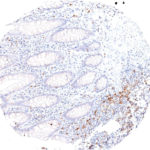
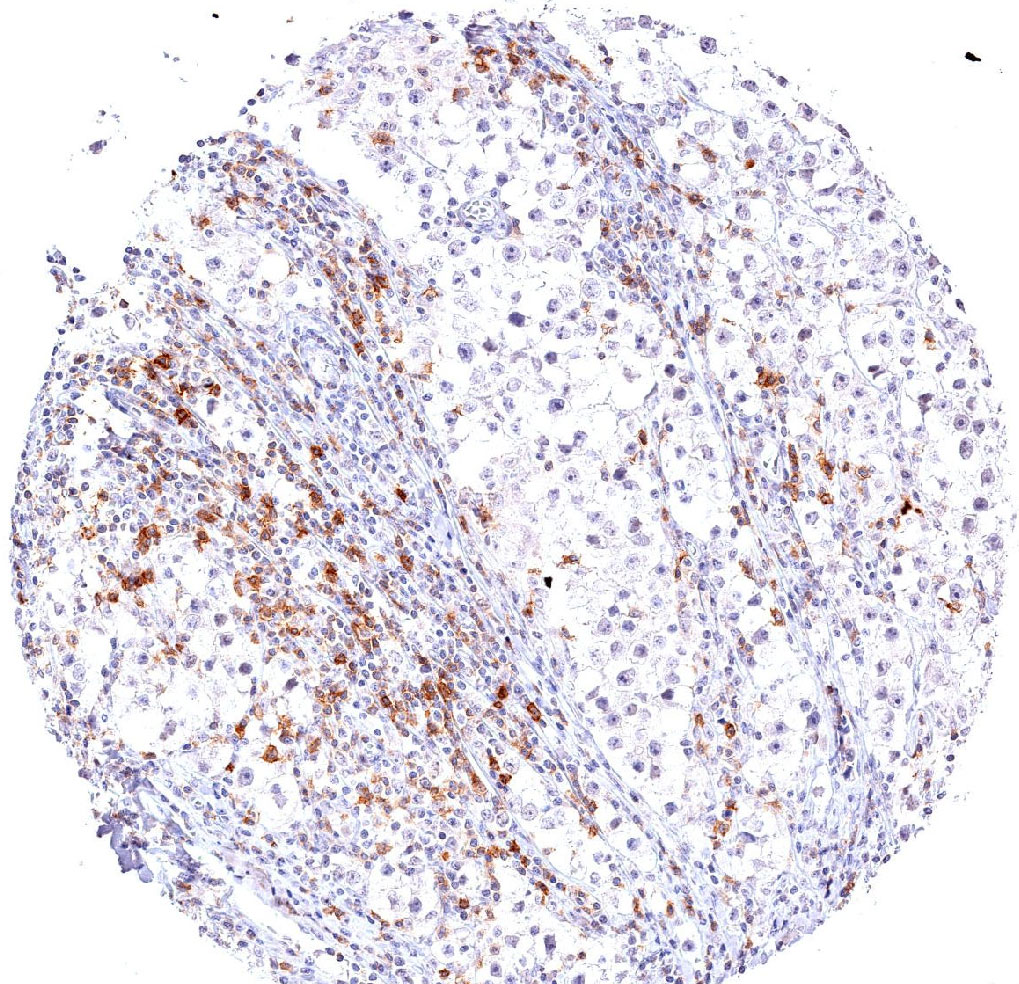
TIGIT im Immuncheckpoint
Immuncheckpoint-Proteine bieten vielversprechende Ansätze für die Krebstheraphie. Krebszellen können zusammen mit Zellen aus der Mikroumgebung des Tumors die körpereigene Immunabwehr unterdrücken, indem sie lokal immunhemmende Komponeten heraufregeln. Der TIGIT-Signalweg spielt eine wichtige Rolle bei der Hemmung der Immunantwort und verknüpft verschiedene Immuncheckpoints. Die neuen immunotherapeutische Ansätze setzen daher auf die Hemmung der TIGIT-Aktivität, insbesondere in einer Kombination mit weiteren Immuncheckpoint-Hemmern.
Der Immunrezeptor TIGIT (T-Zell-Immunrezeptor mit Ig- und ITIM-Domänen) gehört zur Familie der Poliovirus-Rezeptoren (PVR). TIGIT wird auf NK-Zellen, regulatorischen T-Zellen, follikulären T-Helferzellen, Gedächtnis-CD4 + T-Zellen und CD8 + T-Zellen exprimiert, aber nicht auf B-Zellen oder naiven CD4 + T-Zellen. TIGIT wirkt dabei auf einem hochkomplexen Weg als inhibitorischer Immuncheckpoint sowohl auf T-Zellen als auch auf natürliche Killerzellen (NK). Zu den bekannten Liganden für TIGIT gehören CD155 und CD112. Darüberhinaus interagiert das TIGIT / CD155 / CD112-Netzwerk mit weiteren Checkpoint-Reglern.
Studien an Entzündungs- und Krebsmodellen zeigen, dass T-Zellen die TIGIT-Expression hochregulieren können. Auch die TIGIT-Liganden CD155 und CD112 werden auf dendritischen Zellen und Makrophagen bei verschiedenen Krebsarten stark exprimiert. Darüberhinaus korreliert die TIGIT-Expression stark mit der Expression anderer co-inhibitorischer Moleküle, einschließlich PD-1. Zusätzlich zur direkten Hemmung zytotoxischer T-Zellen kann TIGIT weitere Immunzellen beeinflussen, um eine immunsupressive Tumorumgebung zu stimulieren. TIGIT bindet beispielsweise auf der Oberfläche dendritischer Zellen CD155 und hat einen Einfluß auf die Aktivität von NK-Zellen. Es sind zahlreiche Arzneimittel in der Entwicklung, die die Aktivität von TIGIT hemmen.
Antikörperklon TG1
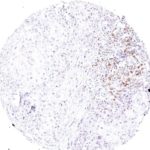
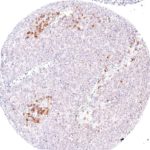
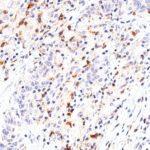
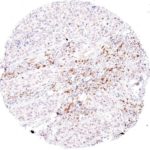
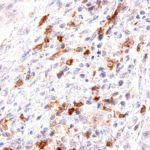
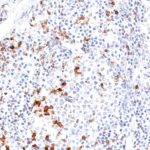
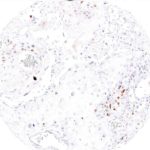
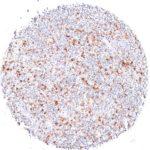
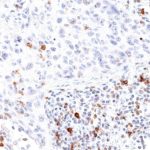
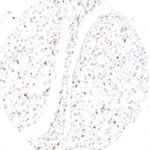
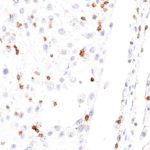
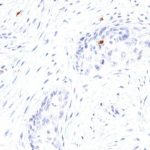
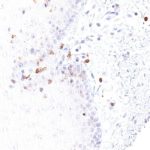
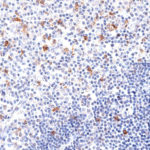
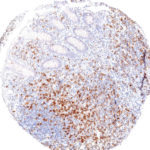
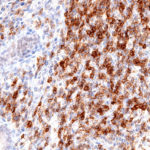
Der Klon TG1 ist der erste monoklonale Antikörper für den immunhistochemischen (IHC) Nachweis von TIGIT (T-Zell-Immunrezeptor mit Ig- und ITIM-Domänen) in standardmäßig Formalin-fixierten und Paraffin-eingebetteten Gewebeproben. TG1 wurde auf zahlreichen Tumoren für den IHC-Nachweis TIGIT-positiver T-Zellen in der Tumormikroumgebung validiert. Histologische Studien mit TG1 an Tumorgeweben können wertvolle Hinweise für die klinische Forschung und für therapeutische Interventionen liefern, die am TIGIT-Tumorimmunologie-Checkpoint ansetzen.
Neu – Klon TG2
Der neu entwickelte Klon TG2 hat eine höhere Affinität zum Zielprotein TIGIT und zeigt in IHC-Vergleichstudien mit Klon TG1 auf Tonsille eine stärkere Färbeintensität.
- Höhere Affinität im Vergleich zu dem Vorgänger TG1 (auf Tonsille getestet)
- Stärkeres Färbesignal
- Höhere Verdünnbarkeit
1.
Li W et al. Expression of the immune checkpoint receptor TIGIT in Hodgkin’s lymphoma. BMC Cancer 2018, 18: 1209.
doi.org/10.1186/s12885-018-5111-1
2.
Blessin NC et al. Patterns of TIGIT expression in normal lymphatic tissue, inflammation and cancer. Disease Markers 2019, Jan 10;2019:5160565. eCollection 2019.
doi.org/10.1155/2019/5160565
3.
Hinsch A et al. et al. Expression of the immune checkpoint receptor TIGIT in seminoma. Oncol Lett. 2019, 18: 1497–1502.
doi: 10.3892/ol.2019.10428
4.
Scimeca, M. et al. Programmed death ligand 1 expression in prostate cancer cells is associated with deep changes of the tumor inflammatory infiltrate composition. Urol. Oncol. 2019, 37, 297.e19-297.e31.
doi: 10.1016/j.urolonc.2019.02.013
5.
Annibali, O., Bianchi, A., Grifoni, A. et al. A novel scoring system for TIGIT expression in classic Hodgkin lymphoma. Sci Rep 11, 7059, 2021.
doi: 10.1038/s41598-021-86655-8
6.
Murakami D et al. Prognostic value of CD155/TIGIT expression in patients with colorectal cancer. PLoS ONE 17(3), 2022.
doi.org/10.1371/journal.pone.0265908
7.
Niebel D et al. DNA methylation regulates TIGIT expression within the melanoma microenvironment, is prognostic for overall survival, and predicts progression‑free survival in patients treated with anti‑PD‑1 immunotherapy. Clinical Epigenetics 14:50, 2022.
doi.org/10.1186/s13148-022-01270-2